벌크 시퀀싱 또는 공간 전사체학 데이터의 표현형을 보려면,
종종 조직병리학(또는 전체 슬라이드 이미징) 데이터와 함께 사용됩니다.
본 연구를 시작한 이유는 Harvard Peter Park 연구소에서 정상 조직에 대한 CNV 연구가 활발히 진행되고 있는데, 정상인의 초기 암 표현형으로 여겨지는 CNV가 WSI에서 나타날 수 있는가?이 주제에 대한 공동 작업이 있습니다.
조직병리학에서 CNV를 검출할 수 있는 능력이 어느 정도 있다면 초기 암 표현형으로 간주되는 증식과 같은 영역을 중요한 플라크로 선택할 수 있습니까?다음 질문에 대한 연구입니다.
저는 주로 MIL(Multiple Instance Learning)로 이 문제를 해결하려고 합니다.
이 글을 쓰는 이유는 조직병리학을 다루는 MIL이라는 방법과 향후 연구를 위해 어느 정도 GNN이라는 방법의 가능성과 한계를 정리하기 위함이다.
1. 다중 인스턴스 학습(MIL)
MIL은 레이블이 지정된 데이터에 포함된 여러 데이터를 처리하는 방법입니다.
예: 각 데이터를 나타냅니다.
가방: 여러 인스턴스가 있는 컬렉션
이때 Bag에는 정답에 해당하는 라벨이 부여된다.
여러 가지 경우가 있을 수 있지만 각 Bag이 서로 다른 경우에 존재할 수 있기 때문에 구조화된 데이터만 다루는 접근 방식은 이와 관련된 문제를 일으킬 수 있습니다. 따라서 여러 인스턴스에서 정보를 추출하는 “집합” 접근 방식이 매우 중요한 문제임을 알 수 있습니다.
Histopathology의 MIL이라면 Bag은 WSI를 나타내고 Instance는 그 안에서 패치라고 할 수 있습니다.
WSI가 너무 크기 때문에 한 번에 처리할 수 있는 CNN을 구현하는 것은 거의 불가능하므로 패치에서 기능을 추출하여 통합하는 방법은 오늘날 MIL의 표준 방법으로 간주됩니다.

Harvard Faisal 실험실의 CLAM은 아래에 설명된 바와 같이 최근의 주요 조직병리학적 방법 중 하나입니다.
– CLAM(클러스터 제한 주의 MIL)
루, 밍 Y. 등. “전체 슬라이드 이미지의 데이터 효율적이고 약한 감독 전산 병리학.” 자연의생명공학과 5.6(2021): 555-570.
CLAM은 클러스터 기반 주의 메커니즘을 통해 레이블을 예측하는 데 도움이 되는 “차별적인 관심 영역”을 예측하는 방법입니다. 목표는 유사한 특성을 가진 패치를 그룹화하고 클러스터에 따라 가중치를 할당하여 최종 패키지 수준 레이블을 예측하는 것입니다.
아래에 자세히 설명된 클러스터 기반 주의 메커니즘 외에도 밀에 의해 조직병리학을 다루는 논문은 거의 비슷한 작업 흐름을 가지고 있습니다.

매우 큰 WSI 이미지에서 패치가 추출되고 “사전 학습된 CNN”을 통해 기능이 추출됩니다. 물론 이 “사전 학습된 CNN”을 사용하는 방법은 매우 다양할 수 있으며, 이미지넷 레이블인 1000개의 소프트맥스 값을 사용할 수 있으며 특징은 CNN 레이어의 3D 특징을 풀링하여 얻을 수 있습니다.

CLAM 연구에서는 256*256*3 이미지를 입력으로 받고 ResNet50 모델의 세 번째 잔차 블록의 출력에 평균 공간 풀링을 적용하여 1024개의 특징 벡터를 얻습니다.
여기서 MIL의 단점이 노출되는데, 특징 추출 과정에서 사용되는 가중치가 end-to-end 방식으로 미세 조정되지 않기 때문에 imageNet 사전 훈련된 모델이 병리학적 이미지를 잘 분해할 수 있다고 가정할 필요가 있습니다. 이 문제를 해결하기 위해 semi-supervision과 같은 방법을 적용하여 훈련 중 특징의 해상도를 향상시킵니다(각 인스턴스는 약하게 감독되기 때문에 서로 다른 특징을 가진 인스턴스를 구별하는 것이 bag-label 학습에 도움이 될 것이라고 가정)).
모든 MIL 방법이 유사하므로 각 인스턴스의 가중치를 추정할 수 있는 방법을 사용하여 최종적으로 가시화된다.
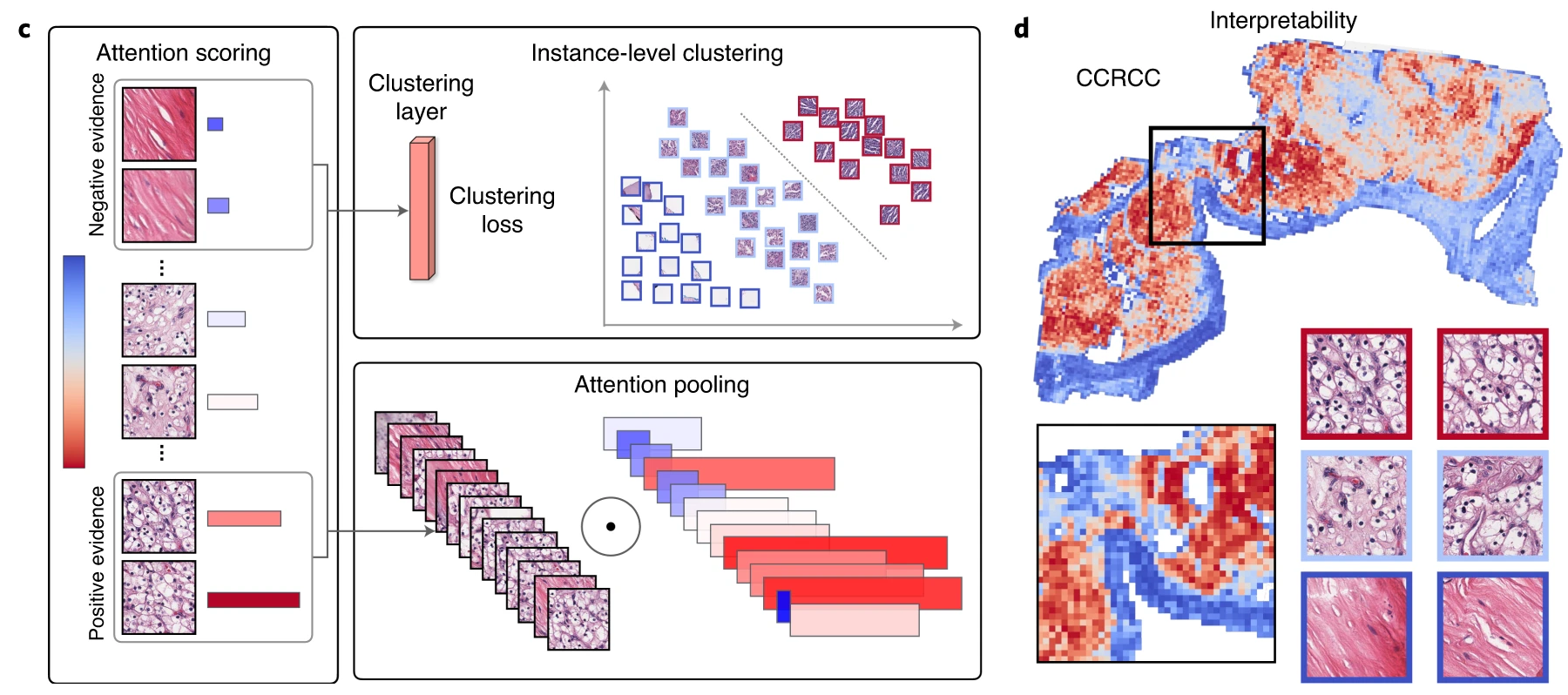
어텐션 점수를 통해 히트맵을 그리는 것은 매우 간단하지만 어떻게 하면 어텐션을 얻을 수 있을까요? 기본적으로 이 문제를 해결하는 아래 문서를 따를 수 있습니다. (2018년도 그런 논문 하나.. 정말 좋은 것 같아요)
Ilse, Maximilian, Jakub Tomczak 및 Max Welling. “주의 기반 심층 다중 인스턴스 학습.” 기계 학습에 관한 국제 회의. PMLR, 2018. (이 기사에 제시된 코드는 정말 이해하기 쉽습니다. )
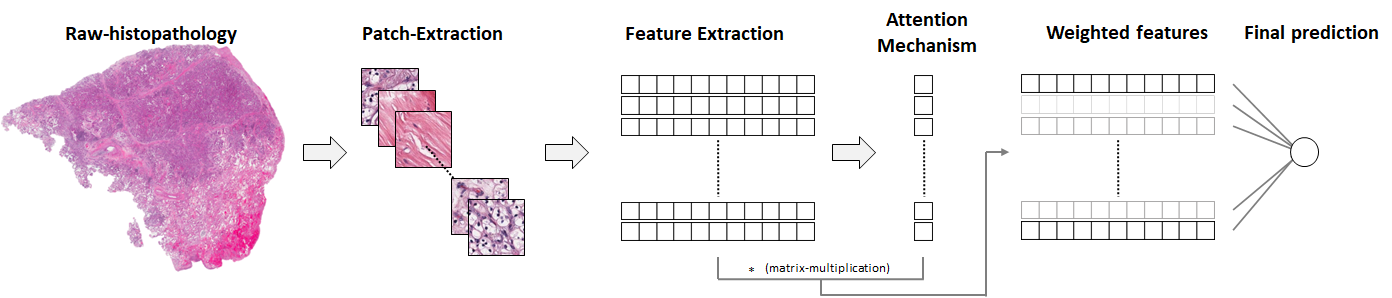
내가 그렸다”주의 기반 심층 다중 인스턴스 학습. “이것이 논문의 Worflow 체계입니다.
위의 그림에서 ‘Feature Extraction’과 ‘Attention mechanism’이라는 두 가지 개념이 가장 중요합니다.

– 기능 추출기
CNN을 사용하는 것은 중간 기능을 선택하는 것입니다. 그러나 CNN 레이어의 출력은 Unit(N) * Matrix(2차원)에 해당하는 3차원 데이터이므로 각 행렬은 ‘Global Average Pooling’과 같은 방법으로 점(0차원)으로 축소되어야 합니다. ‘ . .
즉, 최종 Vector(1d)와 같이 해야 하므로 N개의 행렬로 구성된 CNN의 출력을 풀링해야 한다.
– 주의 메커니즘
선형 회귀의 가장 기본적인 경우(Y = aX + b)에서 입력 X에 대한 Y에 대한 계수 ‘a’의 영향으로 해석할 수 있습니다.
이를 확장하여 다차원 입력 데이터가 들어오는 경우를 생각해보자.
다차원 데이터를 다루는 계층은 선형 회귀와 같이 즉시 해석 가능한 계수(가중치, 매개변수)를 가질 수 없기 때문에 다차원 입력 Xk를 0차원 포인트 값으로 변환하는 네트워크가 생성되었습니다.
네트워크의 출력과 입력 Xk 간의 곱셈을 통해 데이터 자체에 주의를 기울이는 네트워크를 상상할 수 있습니다.
위의 상위 워크플로우 시나리오에서 (“Attention-based deep multi-instance learning workflow scheme”) 추출된 특징 벡터(각 Xk)에 어텐션 가중치를 곱하여 다음 레이어로 넘기는 것을 볼 수 있다.
학습이 원활하게 진행됨에 따라 출력에 영향을 미치지 않는(fade to white) 입력 Xk는 작은 가중치로 걸러내고, 출력에 좋은 영향을 미치는(fade to black) Xk는 높은 가중치를 두고 가중치를 필터링합니다. . 이를 통해 다중 입력의 중요성을 얻을 수 있습니다. 이것이 주목의 개념입니다.
요약, 다중 인스턴스 학습
MIL 기술을 요약하면, 조직병리학적 데이터를 처리하기 위해 현재 사용되는 MIL은 다음과 같다.
특징을 다차원 입력으로 추출하고 매트릭스(행: 패치, 열: 추출된 특징) 형태의 데이터를 사용하여 병리학적 수준 레이블을 일치시킵니다.
어려워보이지만 간단한 매트릭스 데이터로 레이블을 맞추는 것 이상은 아닌 것 같습니다. 여기서 문제는 주의 메커니즘을 사용하여 어떤 패치(행)가 중요한지 추론하고, 조직병리학적으로 중요한 영역을 확인하고, 이를 병리학적 지식과 연결하는 것입니다.
MIL 한계
경계가 명확합니다. 특징 추출기가 병리 이미지의 질감 특징을 잘 구분할 수 있다고 가정해야 합니다. 과거 머신러닝 시대처럼 특징 추출과 모델링이 분리됐을 때 최적의 모델 학습이 어렵다는 뜻임을 알 수 있다.
MIL 논문을 읽으면 암 종류 분류, 암과 정상 분류, 전이 조직 찾기 등 병리학 전문지식이 없는 일반인도 충분히 할 수 있는 일이다. 즉 아주 쉬운 것만이 표적이 되어 기술이 나오고 있다는 뜻이다.
성별을 예측하기 위해 유방 조직에서 MIL을 사용하는 것은 초등학생도 매우 빠르게 할 수 있는 것입니다. 내 경험상 MIL은 이런 종류의 작업에 아주 아주 좋습니다. 그러나 식도와 같이 성별 특성이 명확하지 않은 병리학적 영상에서는 MIL을 사용하여 구분하기 어렵다. 전문가들은 상피, 결합조직의 정도 등 공간정보를 통해 대략적인 추측은 할 수 있지만, 현재 기술로는 MIL을 통해 예측하기가 쉽지 않다. 이는 MIL에서 사용되는 특징 추출기가 식도 샘플에 대한 수출병리학자의 성차별적 근거를 인지하지 못하고 단순 텍스처 특징을 추출하기 때문이다.
심층 학습을 위해 광범위한 조직 질감 특징을 사용하여 유전적 돌연변이, CNV 등을 모호하게 쳐서 0.7 정도의 AUC를 달성하는 논문이 많이 있지만 이는 시기상조인 것 같습니다. AUC가 심지어 0.7인 이유는 개인적으로 KRAS와 다른 유전자 변이는 texture feature에서 발견되지 않지만 암의 단계와 중증도는 KRAS 변이와 다른 유전적 사건과 유의미한 연관(또는 상관관계)이 있기 때문입니다. .
2. 그래프 신경망(GNN)
그래프 구조 데이터는 “엔티티” 간의 복잡한 관계 또는 상호 작용을 설명하는 데 매우 유용합니다. 그래프 자체는 엔터티를 노드로 정의하고 관계를 “에지”로 정의하여 사용할 수 있습니다.
병리학적 이미지를 처리하기 위해 GNN에서 가장 기본적인 용어를 정의해 보자.
노드: 조직 내의 생물학적 구조(세포)
에지: 노드 간의 관계(공간적 근접성)
그래프: 노드 집합과 그래프
이러한 그래프 구조를 신경망에 넣기 위해서는 행렬 구조로 바꿔야 한다.
– 인접 행렬: 노드 간의 관계에 대한 데이터
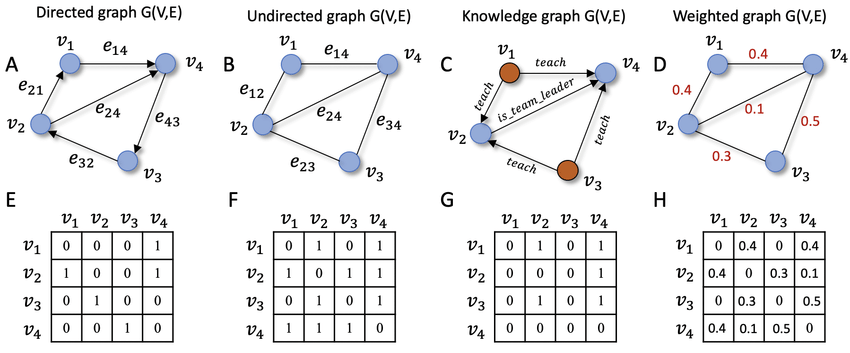
인접 행렬은 각 노드 간의 관계에 대한 데이터를 포함할 수 있습니다.
(행: 노드, 열: 노드 간의 관계)
각 노드에는 특성도 있을 수 있습니다.
(행: 노드, 열: 기능)
예를 들어 기본 GNN은 두 개의 입력을 받습니다.
(인접 행렬, 특징 행렬)
* 전파 규칙
이제 위의 E에 해당하는 인접 행렬에는 4개의 노드 간의 관계를 나타내는 데이터가 있습니다.
따라서 두 개의 기능이 있는 기능 매트릭스가 있다고 가정합니다.
전파는 인접 행렬과 기능 행렬의 곱으로 표현할 수 있습니다.
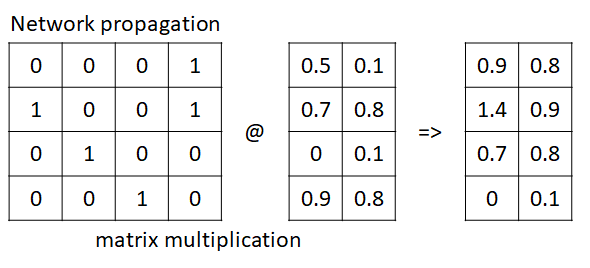
위의 행렬 곱셈을 생각해 봅시다.
첫 번째 노드는 0, 0, 0, 1~4 노드의 관계를 가지며 각 노드는 중간 행렬을 특징으로 합니다.
그들 사이의 행렬 곱셈은,
((0*0.5, 0*0.7, 0*0.0, 1*0.9),
(0*0.1, 0*0.8, 0*0.1, 1*0.8))
사실 잘 생각해보면 첫 번째 노드에 연결된 모든 노드의 특징을 컨볼루션으로 생각할 수 있습니다.
물론 네트워크의 가중치에 따라 가중치는 곱해지고 더해지는 연결된 노드의 특성임을 알 수 있습니다.
따라서 각 노드의 특성은 연결된 노드 사이의 특성에 대한 정보 전파로 간주할 수 있습니다.
수식으로 표현하면 다음과 같습니다.
에프(ⁱ, ㅏ) = σ(아ⁱ와ⁱ)
H: 기능 맵
A: 인접 행렬
W: 가중치 행렬
물론 위의 예는 단순한 전파이며, 그들 사이에 가중치가 주어지면 이것은 그래프 신경망이 됩니다.
전파 과정에서 스킵 연결을 추가하여 ResNet과 같은 특성을 가질 수 있으며 주의 메커니즘도 이 과정에서 부여할 수 있습니다.
이와 같이 노드 간의 관계인 인접 행렬과 노드의 특징을 나타내는 특징 행렬의 두 가지 입력을 통해 원하는 전체 슬라이드 수준에서 학습을 시도할 수 있습니다.
아래 이미지는 AI를 사용하여 병리 이미지를 분석하는 유명 스타트업인 PathAI의 웨비나 프레젠테이션에서 인용한 것입니다.
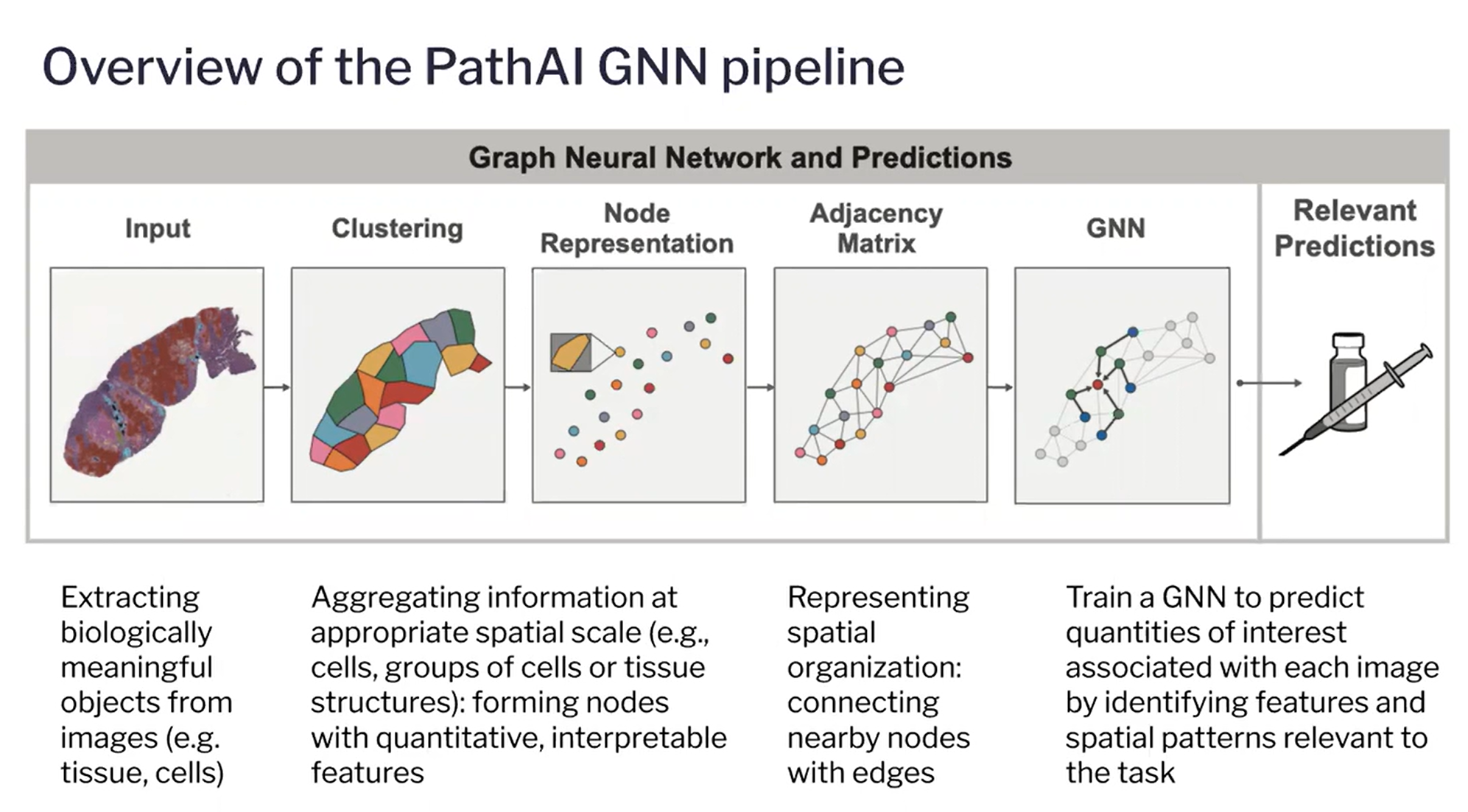
조직병리학에서 세포 및 조직 데이터를 추출합니다.
그러나 노드가 너무 많으면 하드웨어의 한계로 계산이 불가능하므로 클러스터링을 통해 공간정보를 제대로 얻는다.
그런 다음 GNN을 통해 알고 싶은 레이블을 예측하는 방법을 배울 수 있습니다.
##
전체 슬라이드 이미지로 알려진 매우 큰 이미지를 처리하기 위한 두 가지 기술을 살펴보았습니다.
다중 인스턴스 학습 및 그래프 신경망,
두 기술 모두 연구 수준에서 여전히 사용되며 각각 장단점이 있습니다.
앞으로 많은 연구에 활용될 것 같습니다.